
- Autor Miles Stephen stephen@answers-science.com.
- Public 2023-12-15 23:32.
- Zuletzt bearbeitet 2025-06-01 05:03.
Was ist der Unterschied zwischen Mismatch-Reparatur und Nukleotidexzisionsreparatur ? In Reparatur von Fehlanpassungen , einer Nukleotid ersetzt wird, während in Nukleotidexzisionsreparatur mehrere Nukleotide werden ersetzt. In Reparatur von Fehlanpassungen , mehrere Nukleotide ersetzt werden, während in Nukleotidexzisionsreparatur es ist nur eins.
Was ist hier die korrekte Definition der Nukleotidexzisionsreparatur?
Nukleotidexzisionsreparatur ist ein DNA-Reparatur Mechanismus. Nukleotidexzisionsreparatur (NER) ist ein besonders wichtiges Exzision Mechanismus, der entfernt DNA Schäden durch ultraviolettes Licht (UV). UV DNA Beschädigung führt zu sperrig DNA Addukte - diese Addukte sind meist Thymindimere und 6, 4-Photoprodukte.
Zweitens, was passiert während der Nukleotidexzisionsreparatur? Bei der Nukleotidexzisionsreparatur (NER), beschädigte Basen werden innerhalb einer Reihe von Nukleotide , und durch DNA ersetzt, wie durch den unbeschädigten Matrizenstrang angewiesen. Dies Reparatur System wird verwendet, um durch UV-Strahlung gebildete Pyrimidin-Dimere sowie Nukleotide durch sperrige chemische Addukte modifiziert.
In ähnlicher Weise kann man fragen, welche Art von DNA-Mutation üblicherweise durch Nukleotidexzisionsreparatur repariert wird?
Nukleotidexzisionsreparatur ist die primäre Reparatur System für sperrige DNA Addukte wie Cyclobutan-Pyrimidin-Dimer (PyrPyr), (6-4)-Photoprodukt, Benzo[a]pyren-Guanin-Addukt, Acetylaminofluoren-Guanin (AAF-G) und Cisplatin-d(GpG)-Diaddukt.
Was ist das Endergebnis der Basenexzisionsreparatur?
Basenexzisionsreparatur (BER) korrigiert klein Base Läsionen, die die DNA Helixstruktur. Solche Schäden typischerweise Ergebnisse B. durch Desaminierung, Oxidation oder Methylierung (Abb. Lindahl suchte nach einer Enzymaktivität, die auf genomisches Uracil resultierende durch Cytosin-Deaminierung.
Empfohlen:
Was ist der Unterschied zwischen der P-Generation f1-Generation und der f2-Generation?

P bedeutet Elterngeneration und sie sind die einzigen reinen Pflanzen, F1 bedeutet erste Generation und sie alle sind Hybriden, die das dominante Merkmal aufweisen, und F2 bedeutet zweite Generation, die die Enkel von P sind. Wenn ein Individuum ein dominantes Allel hat, wird es zeigen
Was ist der Unterschied zwischen der Kontinentaldrift-Meeresbodenspreizung und der Plattentektonik?

Die Kontinentaldrifttheorie wurde entwickelt, um zu erklären, wie die Ausbreitung des Meeresbodens die Kontinente beeinflussen muss. Die Theorie der Plattentektonik wurde entwickelt, um die Lage von ozeanischen Gräben, Vulkanen und die Lage verschiedener Arten von Erdbeben zu erklären
Was ist der Unterschied zwischen der Masse eines Protons und der Masse eines Elektrons?
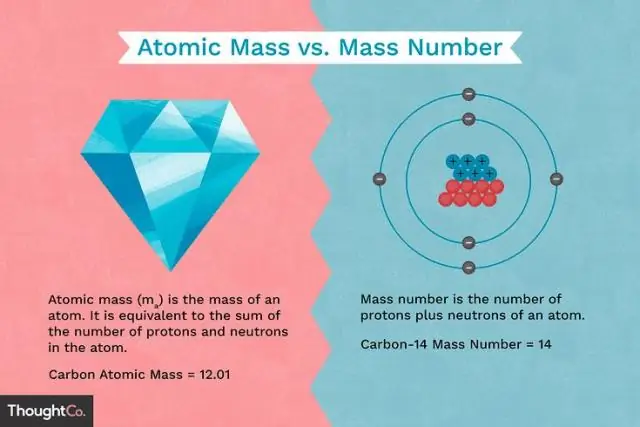
Protonen und Neutronen haben ungefähr die gleiche Masse, aber sie sind beide viel massiver als Elektronen (ungefähr 2.000 Mal so massiv wie ein Elektron). Die positive Ladung eines Protons ist betragsmäßig gleich der negativen Ladung eines Elektrons
Was ist der Unterschied zwischen der Arrhenius-Definition und der Brønsted-Lowry-Definition von Säuren und Basen?

Der Unterschied zwischen den drei Theorien besteht darin, dass die Arrhenius-Theorie besagt, dass die Säuren immer H+ und die Basen immer OH- enthalten. Während das Bronsted-Lowry-Modell behauptet, dass Säuren Protonendonatoren und Pronakzeptoren sind, müssen Basen kein OH- enthalten, also geben Säuren ein Proton an Wasser ab und bilden H3O+
Was ist der Unterschied zwischen der räumlichen Perspektive und der ökologischen Perspektive in der Geographie?
Was ist der Unterschied zwischen der ökologischen Perspektive und der räumlichen Perspektive in der Geographie? die räumliche Perspektive ist, wo etwas passiert oder wo etwas ist. die ökologische Perspektive ist die Wechselwirkung zwischen den Dingen in der Umwelt
